Le mutazioni - Classificazione in base agli effetti - mutazioni con perdita di funzione
In un Post Precedente - Le Mutazioni - definizione e agenti mutageni abbiamo trattato la definizione di mutazione, i principali agenti mutageni e altre cause delle mutazioni.In un secondo post - Le Mutazioni - I tipi di mutazione abbiamo classificato le mutazioni in base al tipo e all'estensione: Mutazioni Puntiformi, Genomiche e Cromosomiche.
Nell'ultimo post abbiamo preso in esame le mutazioni neutre e silenti. Mi baserò su quello scritto nel post per classificare le mutazioni con perdita di funzione
1- Le Mutazioni Missenso.
Nelle mutazioni missenso, la mutazione in uno o più codoni cambia il "senso" dell'informazione contenuta in esso. Uno o più aminoacidi della proteina vengono sostituiti da uno o più aminoacidi che hanno gruppi R di diversa specificità e quindi non possono adempiere alla stessa funzione dell'aminoacido originali.Quindi le sostituzioni Missenso si distinguono da quelle neutre non tanto per il tipo di mutazione quanto per il significato della stessa.
Le mutazioni missenso spesso provocano effetti deleteri anche per la sostituzione di un singolo aminoacido: sostituzione che può alterare la struttura e la funzione di una proteina.
Abbiamo visto nel post precedente che l'ingresso di una Glicina o di una Prolina in un alfa-elica la destabilizza, che la sostituzione di un aminoacido polare con uno non-polare oppure uno acido con uno basico possono essere pericolose, soprattutto se avvengono in un punto cruciale della struttura della proteina, quale ad esempio un sito attivo o allosterico di un enzima.
Abbiamo anche detto che il codice genetico si è "adattato" per ammortizzare gli effetti di queste mutazioni. Generalmente sono più pericolose le Transversioni, sostituzione di Purina con Pirimidina e viceversa, che le Transizioni, sostituzione di Purina con Purina o Pirimidina con Pirimidina.
Quindi le Transizioni, che statisticamente sono più frequenti spesso sostituiscono un aminoacido con uno simile, o per la ridondanza del codice con lo stesso.
(non è una regola assoluta: dalla lettura del codice si vede che anche una transizione può cambiare il tipo di aminoacido ma in un numero minore di casi)Vediamo un esempio:
L'Emoglobina e l'Anemia Falciforme.
Probabilmente l'esempio più famoso in cui basta la sostituzione di una singola base nella sequenza per alterare drammaticamente la funzionalità della proteina.
Emoglobina:
Proteina con struttura quaternaria e gruppi prostetici che nella forma adulta consta di 4 catene polipeptidiche: due catene Alfa identiche e due catene Beta identiche. Ognuna di queste catene trasporta l'Eme il gruppo prostetico che contiene il Ferro ed è in grado di legare reversibilmente l'Ossigeno.
L'Emoglobina si carica di Ossigeno nei polmoni e lo rilascia nei tessuti per alimentare i mitocondri e la respirazione cellulare. Qualsiasi malfunzionamento di questo sistema porta a malattie chiamate Anemie, che possono avere molteplici cause.
L'Emoglobina non è sciolta nel plasma ma è contenuta all'interno dei globuli rossi che, nella forma matura sono a tutti gli effetti dei "sacchetti pieni di emoglobina", hanno addirittura eliminato il nucleo cellulare per avere più spazio.
Ovviamente i Globuli Rossi, privi di nucleo, hanno un periodo di vita limitato, attorno ai 120 giorni.
Vengono continuamente demoliti nel fegato e continuamente il midollo osseo ne forma di nuovi.
In un singolo millimetro cubo di sangue ci sono dai 4 ai 6 milioni di Globuli Rossi!
L'Anemia Falciforme:
L'anemia falciforme è una malattia in cui la forma, la durata di vita e la funzionalità dei globuli rossi è estremamente alterata.
Quando si caricano di Ossigeno hanno la normale forma Biconcava, quando invece restituiscono l'Ossigeno ai tessuti si deformano e assumono una forma a Falce (Sickle in inglese), da qui il nome dell'anemia.
Questo cambiamento di forma altera il flusso dei globuli rossi nel torrente sanguigno a livello dei capillari, causando "tappi" che possono provocare il blocco della circolazione e ischemie e soprattutto altera la vitalità dei globuli rossi che si riduce di 10 volte, costringendo Midollo Osseo e Fegato a un carico di lavoro immenso per la loro sostituzione. L'organismo, a seconda della gravità della forma anemica soffre di un continuo stato di ipossia (e molti altri problemi che esulano da questa trattazione)
Tutti questi problemi, che si ripercuotono in tutto l'organismo, sono causati da una SINGOLA sostituzione di una base nel gene codificante per la catena Beta dell'emoglobina.
Si tratta di una transversione di Adenina in Timina, nel sesto codone della proteina.
Come risultato un codone GAG cambia in GUG.
GAG codifica per un aminocido carico, l'acido Glutammico mentre il GUG codifica per un aminoacido neutro la Valina.
La Valina, che sostituisce l'acido glutammico nella forma mutata, è in grado di formare dei ponti idrofobici con le catene di altre emogobine adiacenti, quando la proteina è scarica di ossigeno.
Fonte: Semantic ScholarSi formano quindi delle vere e proprie fibrille compattate di emoglobina che alterano la struttura del globulo rosso.
Fonte dell'immagine: biochemistry course
Vediamo un altro esempio:
La Fenilalanina Idrossilasi e la Fenilchetonuria
Fenilalanina Idrossilasi:
La Fenilananina è un aminoacido essenziale.Il che vuol dire che deve essere assunto con la dieta in quanto non siamo in grado di produrlo.
La Fenilalanina è uno dei 20 aminoacidi che entrano a far parte delle proteine.
Quindi è una molecola INDISPENSABILE per il nostro organismo.
Però l'eccesso di aminoacido presente nel sangue causa una malattia chiamata Fenilchetonuria (PKU).
Normalmente l'eccesso di Fenilananina non utilizzata entra in una via catabolica che ha come tappa chiave la sua trasformazione in Tirosina, un altro aminoacido che può essere eliminato più facilmente.
L'Enzima che si occupa di questa tappa metabolica è la Fenilalanina Idrossilasi (PAH): così chiamato in quanto aggiunge un gruppo -OH all'anello aromatico della Fenilalanina trasformandola in Tirosina.
Quando l'enzima è poco o non funzionale, la via degradativa dell'aminoacido sceglie una strada diversa che lo trasforma in Acido Fenilpiruvico.
L'accumulo progressivo di derivati catabolici di acido Fenilpiruvico è tossico e causa problemi soprattutto al SNC con grave disabilità intellettiva.
Senza entrare nel dettaglio della malattia, l'enzima PAH può essere soggetto a numerose mutazioni di diverso tipo. Una delle più importanti, come nel caso dell'anemia falciforme, è una singola sostituzione aminoacidica. Nella posizione 408 delle proteina un residuo di Arginina, aminoacido carico positivamente, viene sostituito da uno di triptofano, aminoacido aromatico neutro: p.Arg408Trp.
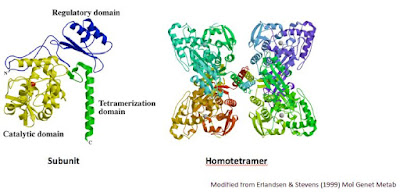
Questa, come la maggior parte delle mutazioni missenso della proteina, cade all'interno del dominio catalitico dell'enzima. Basta questa sostituzione per compromettere la funzionalità enzimatica.
La mutazione è una transizione C-->T nella prima posizione del codone che cambia da CGG a TGG.
Fonte: Loss of Function in Phenylketonuria Is Caused by Impaired Molecular Motions and Conformational Instability
Questa mutazione cade proprio all'estremità del sito catalitico. Non coivolge direttamente l'attività enzimatica ma compromette il corretto avvolgimento della proteina a formare la struttura terziaria e quaternaria.
Anche PAH, come l'emoglobina del precendente esempio è un tretramero. A differenza dell'Emoglobina che è costituita da due catene differenti, la Fenilalanina Idrossilasi è composta da 4 catene uguali che si uniscono tramite l'estremità C-terminale della molecola.
Fonte: PKU academy
Nei pazienti con questa mutazione missenso la Proteina non è in grado di avvolgersi correttamente e forma delle masse prive di attività enzimatica.
2- Le Mutazioni Non-senso
In una mutazione non-senso la proteina ha sempre lo stesso triste destino. Sebbene questo tipo di mutazioni non alterino la funzionalità dell'mRNA hanno un effetto drastico sulla proteina:
Introducono un codone di STOP prima del previsto.
Il risultato è sempre lo stesso: UNA PROTEINA TRONCA.
Ora, in alcuni casi, se il codone di STOP extra si presenta verso la fine della proteina, nell'estremità C-terminale, la proteina potrebbe anche conservare la sua funzione, se invece si presenta precocemente, si ha un prodotto proteico privo di alcune funzionalità e semplicemente non in grado di avvolgersi in una corretta struttura terziaria.
Quindi le mutazioni non-senso raramente conservano una funzionalità.
I codoni di STOP sono, ricordiamolo UAA, UAG e UGA:
Per cui non tutti gli aminoacidi possono essere coinvolti in questo tipo di mutazione.
se guardiamo il codice genetico:
Arginina CGA, AGA, 2 codoni su 6
CGU, CGC, CGA, CGG, AGA, AGG
Cisteina UGU UGA 2 su 2
UGU, UGC
Glutammina CAA, CAG tutti e due i codoni
CAA, CAG
acido Glutammico GAA, GAG tutti e due i codoni
GAA, GAG
Glicina GGA un codone su 4
GGU, GGC, GGA, GGG
Leucina UUA, UUG 2 codoni su 6
UUA, UUG, CUU, CUC, CUA, CUG
Lisina AAA, AAG tutti e due i codoni
AAA, AAG
Triptofano UGG l'unico codone
Tirosina UAU, UAC tutti e due i codoni
UAU, UAC
Se non ne ho dimenticato nessuno.
Ora, sono un mucchio di punti di mutazione. Però ci sono diverse possibilità di venirne fuori con meno danno:
1- Se la mutazione è una transversione è rara.
2- Se la mutazione ricade nella terza posizione del codone, a volte i t-RNA possono comunque agganciare il codone solo tramite le prime due basi. Quindi anche se in piccola percentuale una certa quantità di proteina normale viene prodotta nei ribosomi.
3- A volte i codoni che portano allo STOP sono meno frequenti. Ma non è una regola generale.
Anche il troppo stroppia MUTAZIONI DI ALLUNGAMENTO:
Può succedere anche il contrario!
Un codone di STOP muta in un codone codificante uno degli aminoacidi sopra citati.
La Proteina non sarà TRONCA ma ALLUNGATA:
Anche se questo può in alcuni casi non essere dannoso, bisogna considerare che alla proteina vengono aggiunti degli aminoacidi casuali che ne possono destabilizzare la struttura o ostacolarne il movimento e la processazione nel reticolo endoplasmatico ruvido e nell'apparato di Golgi.
Oppure semplicemente la proteina benché abbastanza funzionante potrebbe essere riconosciuta dal sistema dell'Ubiquitina e essere demolita.
3- Microdelezioni senza sfasamento del Modulo di Lettura
Le sequenze codificanti nel DNA e nell'mRNA maturo non hanno segni di interpunzione.I codoni si succedono uno dopo l'altro e i ribosomi agganciano l'aminoacido corrispettivo.
Ogni codone consta di una sequenza di 3 basi, come abbiamo detto nei post precedenti.
Ogni microinserzione o microdelezione che mi aggiunge 3 nucleotidi (o multipli di 3), oppure mi taglia via 3 nucleotidi (o multipli di 3), non sposta il modulo di lettura.
La mutazione resta confinata in quella posizione senza conseguenze per gli aminoacidi successivi.
Invece inserzioni o delezioni di un numero variabile di nucleotidi che non sia 3 o multiplo di 3 cambiano TUTTI gli aminoacidi che seguono la mutazione (come vedremo poi).
Si potrebbe quindi supporre che l'aggiunta o la sottrazione di tre nucleotidi non comporti grossi problemi per la proteina.Questo a volte è vero, ma bisogna ricordare che vale la stessa regola delle mutazioni missenso: se quell'aminoacido è un aminoacido chiave per la funzionalità della proteina le conseguenze possono essere drammatiche, come abbiamo visto sopra per la talassemia e la fenilchetonuria.
- è il caso di una delle mutazioni che causano la fibrosi cistica...
Vediamo un esempio:
CFTR e la Fibrosi Cistica
CFTR (Regolatore della conduttanza Transmembrana associato alla Fibrosi Cistica)
Si tratta di una imponente proteina integrale della membrana plasmatica che svolge un ruolo fondamentale nella regolazione del flusso del cloro (ione cloruro) verso l'esterno attraverso la membrana delle cellule epiteliali.Un suo malfunzionamento causa problemi più o meno gravi, a seconda della mutazione e della funzionalità residua della proteina, che vanno a colpire diversi organi. Senza addentrarci troppo, l'alterato flusso in uscita del cloro che si trascina dietro anche l'acqua, causa l'accumulo l'indurimento e l'ispessimento dello strato di muco, che normalmente riveste le mucose, provocando il blocco della funzionalità di molti organi, invalidando però soprattutto il sistema respiratorio e il pancreas che non riesce a secernere normalmente gli enzimi pancreatici.
La proteina è molto grande, 1480 aminoacidi, ha molti domini, transmembrana, regolativi, allosterici.
Può sembrare incredibile ma una delle mutazioni più comuni e deleterie di questa proteina è la mancanza di UN SINGOLO AMINOACIDO, per una microdelezione di soli 3 nucleotidi.
La mutazione è la deltaF508:
Questa mutazione comporta la scomparsa di un codone per la Fenilalanina.
I nucleotidi mancanti sono CTT
Dato che i codoni per la Fenilalanina disponibili nel codice genetico sono UUU, UUC nel DNA ovviamente rappresentati da TTT e TTC la mutazione non colpisce la tripletta specifica per la Fenilalanina ma cade a cavallo di due triplette adiacenti.
Fonte: Associazione trentina Fibrosi Cistica
L'aminoacido che precede la Fenilalanina, la Isoleucina, indicata con la sigla Ile nella figura, non viene alterato.
Infatti anche se il codone ATC viene sostituito da ATT, anche quest'ultimo codifica per la Isoleucina.
Quindi alla fine l'unica alterazione è la scomparsa di una Fenilalanina.
Eppure questo aminoacido deve esercitare un ruolo fondamentale nell'avvolgimento della proteina. I geni mutati codificano per proteine CFTR che non sono in grado di avvolgersi correttamente e vengono degradate ancor prima di raggiungere l'apparato di Golgi, dove la proteina normale viene modificata con l'aggiunta di oligosaccaridi e spedita poi nella sua corretta posizione all'interno della membrana cellulare
Nella figura seguente una proteina CFTR con l'ubicazione della mutazione deltaF508 e l'indicazione dei domini della proteina.
4- Le Mutazioni con spostamento del modulo di lettura (frame-shift).
Visto che anche singole sostituzioni aminoacidiche possono causare effetti devastanti sulla funzionalità della proteina, immaginiamo cosa può succedere se si altera una buona parte della proteina.Questo accade ad esempio, come abbiamo detto prima, quando le microinserzioni o le microdelezioni non sono multipli di 3.
In questo caso cambia, dal punto della mutazione in poi, lo schema con cui viene letta la successione di codoni.
Per cui gli aminoacidi che seguono il sito della mutazione sono completamente differenti da quelli che avrebbero dovuto essere inseriti.
Ovviamente una proteina che ha questo tipo di mutazioni ben di rado è funzionale e solo se la mutazione colpisce la parte terminale della proteina non compromettendone la stabilità.
Prendiamo questa sequenza (sono i primi 30 aminoacidi dell'mRNA dell'insulina)
atg gcc ctg tgg atg cgc ctc ctg ccc ctg
M A L W M R L L P L
ctg gcg ctg ctg gcc ctc tgg gga cct gac
L A L L A L W G P D
cca gcc gca gcc ttt gtg aac caa cac ctg
P A A A F V N Q H L
1- inserisco un nucleotide a caso sul quinto codone (una a prima della g)
atg gcc ctg tgg ata gcg cct cct gcc cct
M A L W I A P P A P
gct ggc gct gct ggc cct ctg ggg acc tga
A G A A G P L G T -
ccc agc cgc agc ctt tgt gaa cca aca cct
P S R S L C E P T P
Risultato:
praticamente dopo la mutazione non c'è un aminoacido identico al precedente, è statisticamente molto improbabile in una sequenza così corta, ma succede una cosa abbastanza prevedibile:
Al ventesimo codone, dopo solo 15 aminoacidi mi compare un CODONE DI STOP TGA (UGA)
2- Opero una delezione di due nucleotidi sul settimo codone (gli ultimi due T e C)
atg gcc ctg tgg atg cgc cct gcc cct gct
M A L W M R P A P A
ggc gct gct ggc cct ctg ggg acc tga ccc
G A A G P L G T - P
agc cgc agc ctt tgt gaa cca aca cct
S R S L C E P T P
Ancora una volta mi compare un CODONE DI STOP TGA (UGA)
Comunque ci si provi, una mutazione frame-shift ha sempre queste due conseguenze:
1- Altera la sequenza di aminoacidi successivi alla mutazione. Non c'è ALCUNA possibilità che la nuova sequenza sia funzionale.
2- Prima o poi lo sfasamento fa comparire un codone di stop che interrompe la traduzione della proteina e genera una proteina tronca.
Quindi le mutazioni frame-shift sono compatibili con la funzionalità delle proteine solo se colpiscono le regioni terminali e solo in alcuni casi.
5- Le mutazioni all'interno degli introni
Si potrebbe pensare che le mutazioni all'interno degli introni, dato che non fanno parte della sequenza codificante, possano essere assorbite con più tranquillità.
In molti casi è vero.
Gli INTRONI, sequenze con ruolo regolativo all'interno del gene che sono tagliate via durante il processo di maturazione dell'mRNA prima che venga tradotto nei ribosomi, hanno un tasso di mutazione più alto rispetto agli ESONI, che invece, come abbiamo visto, rischiano ad ogni mutazione di compromettere la funzione della proteina di cui fanno parte.
Ogni gene eucariotico sul DNA è quindi un mosaico di ESONI e INTRONI intervallati.
Ad esempio il gene PAH ha 13 esoni e 12 introni
il gene per il CFTR è spezzettato in addiruttura 27 esoni e 26 introni
il gene per la catena beta dell'emoglobina ha soltanto 3 esoni e 2 introni
Quindi l'mRNA del gene PAH deve effettuare 12 operazioni di splicing per rimuovere gli introni
il gene per il CFTR ben 26 operazioni di splicing per rimuovere gli introni
al gene per catena beta dell'emoglobina bastano due operazioni di splicing per ottenere l'mRNA maturo.
Abbiamo detto in un post precedente che le sequenze all'interno dell'Introne per consentire uno splicing corretto non sono molto lunghe né conservate con rigore.
però...
Può succedere che una mutazione all'interno dell'introne non passi senza problemi in quanto va a colpire un punto nevralgico per effettuare uno splicing corretto.
Lo splicing. in molti introni, introni del gruppo 2, avviene in tre tappe:
1- L'adenina in lilla nella figura aggancia la G della sequenza GT e stacca l'estremità 5' dell'introne dall'esone adiacente. (Introni del gruppo 2)
2- Si forma un cappio o Lariat
3- Questa nuova conformazione viene riconosciuta dallo splisosoma che stacca anche l'estremità 3' dell'introne, riconoscibile per una serie di C/T e soprattutto per l'AG terminale, e contemporaneamente salda le due estremità.
Ci sono quindi tre punti all'interno di ogni introne particolarmente sensibili:
1- L'estremità 5' che determina il sito donatore di splicing (GU - GT nel DNA in figura)
2- l'Adenina (A) nel sito di ramificazione coinvolta nella formazione del cappio e le poche basi limitrofe
3- L'estremità 3' terminale del sito accettore di splicing. (AG)
4- Per quanto riguarda le sequenze esoniche fiancheggianti avevamo già affrontato il problema.
Questi siti sono critici in qualsiasi introne e una variazione può impedire che avvenga uno splicing corretto.
5- A complicare la situazione ci possono essere sequenze che incentivano o disincentivano l'uso di siti di splicing, sia all'interno degli introni che negli esoni, per cui lo studio delle mutazioni che alterano lo splicing è sempre estremamente complesso.
Qualunque sia la conformazione dell'introne, le sue regioni conservate o il meccanismo di splicing, il risultato di una mutazione che lo altera, crea sempre grossi problemi.
Che cosa può succedere a causa di mutazioni che possono alterare il riconoscimento dello splisosoma?
A- L'esone successivo all'introne viene completamente ignorato e viene tagliato via insieme ai due introni che lo circondano.
Avremo una proteina che manca di un ESONE!
Ricordate che evolutivamente questi meccanismi sono alla base dello Splicing alternativo e garantiscono a molte proteine di avere forme e funzioni diverse in tessuti diversi.
Ma se si verificano a causa di una nuova mutazione il destino finale sarà quasi sempre la produzione di una proteina più corta non funzionale.
B- Lo splisosoma cerca comunque di effettuare uno splicing e si aggancia a sequenze simili a quelle di riconoscimento poste o all'interno dell'introne o dell'esone fiancheggiante.
In questo caso avremo dei pezzi in più nell'mRNA, se il nuovo sito di splicing si trova all'interno dell'introne, o pezzi in meno se viene attivata una sequenza all'interno dell'esone.
Queste sequenze simili, normalmente inattive, si chiamano siti di splicing CRIPTICI.
In questo caso non si rispettano i confini degli esoni per cui non si rispetta quasi mai il frame-shift.
Per cui il risultato è praticamente sempre una proteina non funzionante.
VEDI ANCHE:
Le Mutazioni - I Tipi di Mutazione
Le Mutazioni - definizione e agenti mutageni
Le Mutazioni - Classificazione in base agli effetti - le mutazioni neutre
Le Mutazioni - Classificazione in base agli effetti -mutazioni con perdita di funzione
Le Mutazioni - Classificazione in base agli effetti -mutazioni con perdita di funzione










Nessun commento:
Posta un commento